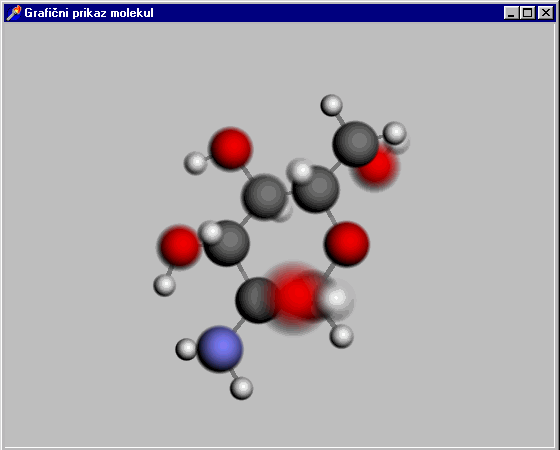
Grafični prikaz molekul

Uvod
Velikokrat si želimo, da bi si določeno molekulo lahko predstavljali v treh dimenzijah, saj si le na ta način lahko molekulo nazorno predstavljamo. To nam koristi na mnogo področjih kot so fizika, kemija, biologija. Namen tega projekta je izdelava programa, ki molekulo prikaze v treh dimenzijah. Program omogoča prikaz molekule iz različnih zornih kotov in bi na različne načine poudari globino, tretjo dimenzijo prostora.
Navodila za uporabo
Program poženemo s Visu.exe. Za tem se pojavita dve okni. Prvo okno kaže model molekule, ki pa je na začetku prazno. Drugo okno je nadzorno, s katerim izbiramo razne nastavitve prikaza, računanja ali pa naložimo novo molekulo.
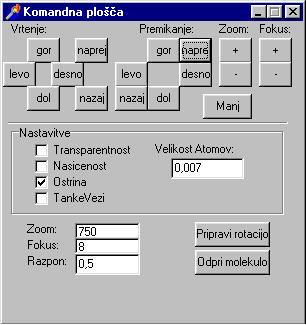
Program nam omogoča vrtenje modela molekule v vseh treh oseh (tako da določimo eulerijeve kote), nastavljanje vezi (lahko so navadne črte ali pa odebeljene pobarvane črte), izbiranje med perspektivo ali pravokotno projekcijo, nastavljanje velikosti atomov, globino poudarka (ostrina pomeni, da so ostri le atomi, ki so
v goriščni razdalji. Nasičenost pomeni, da atomi, ki niso v goriščni razdalji posivijo. Prozornost pa pomeni, da atomi, ki niso v goriščni razdalji delno zbledijo. Pri tem nam pomaga fokus. Fokus je oddaljenost od ostrine do oči. Določimo ga tako da vpišemo v nadzorno okno ali pa v glavnem oknu pritiskamo tipki Page Up ali Page Down. Rotacijo lahko določamo z miško. Levi miškin gumb vrti molekulo, desni pa premika molekulo. Z razponom lahko določamo širino globinskega pasu na katerem vidimo ostro vse atome.
ostrine od uka) fokus določmo tko da ga upišemo v cntrl panl al pa da k main windwu prtiskamo pgup ali pgdn. rotacjo štelamo tko da z mouse1 klikamo & dragamo v prikaznem windowu za mouse2 pa movamo molekulo.
RAZPON .. s tem določamo širino globinskega pasu na katermu vidmo ostro .. ožji k je majn ostrih atomov bomo vidl .. širši k pa je več jih bomo widl .... J
Kako program deluje
Atom
Proceduri za risanje atoma podamo radija a in b ter barvo atoma. Atom potem narišemo tako da od konca radija a pa do kon
ca radija b barva atoma preide v barvo ozadja. Radija a in b ter barva atoma so odvisni od oddaljenosti atoma od očesa.Če sta radija a in b enaka pravimo, da je atom v goriščtni razdlji (označena je z f). Čim bolj je atom izmaknjen iz te razdalje, tem večja je razlika med radijem a in b.
Za barvanje atoma namesto sistema RGB(Red, Green, Blue) uporabljamo sistem HSB, kjer H pomeni barvo atoma, S pomeni nasičenost barve (0=siva, 100=zelo živa barva) in B svetlost barve. S barve atoma pada z oddaljenostjo atoma od očesa.
Da bi dobili najboljše razmerje med H, S, B, a, b, z, in f smo uporabili proceduro KakoizgledaAtom, ki jo bomo po potrebi spremenili. Podamo ji barvo atoma(H, S, B), naravno velikost(r) in trenutno goriščno razdaljo od očesa(f), vrne pa nam
oba radija (a, b) ter barvo(H, S, B).Za boljši 3D prikaz atoma smo uporabili tako imenovani Lambertov kosinusov zakon, ki pravi, da je osvetljenost enote površine je sorazmerna s kosinusom kota med pravokotnico na površino in smerjo padajoče svetlobe.
Vez
Matrike
Matrike nam omogočajo obračanje koordinatnega sistema ali le določene osi sistema. Za vsako os smo naredili posebno matriko s katero lahko vrtimo posamezno os. Če pa želimo vrteti celotni koordinatni sistem pa moramo matrike zmnožiti. Če želimo vrteti določeno točko, jo množimo z zmnožkom matrik.
Perspektiva
S pomočjo vektorjev lahko navidezni koordinatni sistem obrnemo in s tem dosežemo obračanje modela molekul. Ko izračunamo pravokotno projekcijo (x in y koordinate) dobljeno sliko delimo s koordinato z, ki nam na enostaven način pravokotno projekcijo pretvori v perspekti
vo. Namen perspektive je doseči, da je dvakrat bolj oddaljeno telo prikazano dvakrat bližje skupaj. To je očitno obratno sorazmerje z oddaljenostjo.Format HIN
V tej datoteki so podrobno definirani vsi atomi in njihove vezi, ki gradijo določeno molekulo. Da
toteka formatu HIN izgleda takole(za primer benzena):forcefield ambersys 0view 40 0.302419 40 10 1 0 0 0 1 0 0 0 1 0.406355 0.0451501 -40
mol 1 0
atom 1 - C CA - 0 -1.61879 -0.74515 0 3 2 a 6 a 7 s
atom 2 - C CA - 0 -1.61879 0.65485 0 3 1 a 3 a 8 s
atom 3 - C CA - 0 -0.406352 1.35485 0 3 2 a 4 a 9 s
atom 4 - C CA - 0 0.806083 0.654849 0 3 3 a 5 a 10 s
atom 5 - C CA - 0 0.806083 -0.74515 0 3 4 a 6 a 11 s
atom 6 - C CA - 0 -0.406352 -1.44515 0 3 5 a 1 a 12 s
atom 7 - H HC - 0 -2.5541 -1.28515 0 1 1 s
atom 8 - H HC - 0 -2.5541 1.19485 0 1 2 s
atom 9 - H HC - 0 -0.406352 2.43485 0 1 3 s
atom 10 - H HC - 0 1.74139 1.19485 0 1 4 s
atom 11 - H HC - 0 1.74139 -1.28515 0 1 5 s
atom 12 - H HC - 0 -0.406352 -2.52515 0 1 6 s
endmol 1
Podatki do ukaza mol povejo iz kje gledamo molekulo in podobno. Ukaz
mol prične opis posameznega atoma, sledi mu označba molekule. Opis molekule se konča z endmol in označbo molekule. Vmes se posamezni atomi navajajo s stavkom atom takole:atom st – simbol hibridizacija – 0 x y z nvezi atom1 vez1, atom2, vez2… st zaznamuje stevilko atoma, simbol je njegov kemijski simbol(C-ogljik, H-vodik). Za kemijskim simbolom je tip hibridizacije (lahko ignoriramo). x, y, z, so koordinate atoma v prostoru. Nvezi je stevilo vezi, ki jih ima atom, atom1 je atom do katerega je napeljana vez, vez1 pa je tip vezi. Zaključek-